Taxonomía numérica
| ||||||
Taxonomía numérica. Agrupación de unidades taxonómicas por métodos numéricos, la cual ha alcanzado un importante crecimiento que ha permitido el uso de métodos estadísticos multivariados para la clasificación de los recursos genéticos (Sokal y Sneath, 1963).
Sumario
Historia
La clasificación de las especies, tal como se entiende en la actualidad, fue iniciada por Carlos Linneo en su famoso “Sistema Natura”. Linneo describió miles de especies utilizando la nomenclatura binomial, que asignaba a cada viviente el nombre latino con el género y la especie, cumpliendo la función de etiqueta científica, que permitiera situarlo y relacionarlo con las demás especies. El sistema taxonómico de Linneo es una jerarquía organizada en niveles, en la cual las clases disjuntas a cada nivel constituyen las llamadas taxas. Las taxas a un dado nivel constituyen las categorías. Se habla así de las categorías ‘especies’, ‘género’, ‘familia’, ‘orden’, etc. La categoría género, por ejemplo, tiene diversas taxas: los géneros que constituyen a una categoría dada. (Cuadras 1981)
Surgimiento
La taxonomía numérica surgió en los años 60 (Sokal y Sneath 1963; Sneath y Sokal 1973) por la necesidad de ampliar los esquemas tradicionales de la sistemática de los seres vivientes. Esta intenta cuantificar los datos y establecer grupos por la similitud global. Se anota el número de similitudes o características compartidas, y se agrupan en taxones más cercanos aquellas especies con mayor número de similitudes, y en taxones más alejados a medida que el número de similitudes decrece. En líneas generales, la taxonomía numérica intenta construir clasificaciones “naturales”, basadas en la semejanza fenotípica de los individuos (o de las clases), que se valora partiendo de una adecuada elección de un coeficiente de similaridad (Cuadras 1981).
En el análisis de agrupamiento se pueden definir un grupo de procedimientos básicos: análisis de datos, selección de variables, selección de una medida de proximidad a usar como índice de similaridad o disimilaridad entre los objetos, selección del procedimiento de agrupamiento, validación del agrupamiento obtenido y determinar el número conglomerados. Esta secuencia de análisis se puede realizar utilizando las facilidades que brinda un grupo de paquetes definidos en R, que es un lenguaje de programación principalmente orientado al análisis estadístico.
Análisis de datos
El análisis de los datos implica la separación, identificación y medida de variación en un conjunto de variables, este pre-procesamiento es muy amplio, pues abarca desde técnicas de estandarización, normalización hasta posible discretización.
Técnicas de estandarización y normalización
Un problema al que se enfrentan todas las medidas de distancias es que el uso de datos no estandarizados implica inconsistencia entre las soluciones de los conglomerados cuando cambia la escala de las variables. El orden de las similitudes puede cambiar profundamente con sólo un cambio de escala en una de las variables. Debe emplearse, por tanto, la estandarización de las variables para evitar diferentes soluciones, ya que las diferencias existentes entre las variables con puntuaciones muy altas pueden anular las diferencias existentes entre las variables con puntuaciones bajas.
Para resolver este problema suele recomendarse no utilizar las puntuaciones directas de las variables sino las puntuaciones transformadas a escalas del mismo rango.
En la función data.Normalization del paquete clusterSim están implementados 11 tipos de normalizaciones y estandarizaciones. Además, se recomienda incorporar el procedimiento de estandarización que se realiza con la métrica de Gower (1971), la cual estandariza cada variable dentro de un rango de [0,1] (Podani and Schmera 2006), y además permite el análisis de variables mixtas. Esta métrica esta implementada en la función gowdis(), descrita en el paquete FD.
Diferentes técnicas de transformación de los datos
Un patrón es representado a través de atributos. Una transformación de los datos es un proceso por el cual la representación de los patrones es cambiada creando un nuevo atributo. Este nuevo atributo puede ser un subconjunto de los atributos originales. Existen diferentes técnicas de transformación de los datos, por ejemplo el Análisis de Componentes Principales (Smith 2002), PCA por sus siglas en inglés, que es usada para reducir el número de atributos requeridos para representar los datos. Está técnica la podemos encontrar implementada en la función prcomp() del paquete stats, además en la función dudi.pca() del paquete ade4 y en la función PCA() del paquete FactoMineR en lenguaje R.
La Discretización (Dougherty 1995), es otra técnica de transformación de los datos, la cual esta enmarcada en un proceso que divide los valores numéricos continuos, en un conjunto de intervalos que pueden ser considerados como valores de variables categóricas. El proceso de discretización puede mejorar la precisión de los algoritmos de clasificación, además existen numerosas medidas de distancias que trabajan con datos nominales. En el paquete discretization en R podemos encontrar varias funciones de descretización.
Selección de variables
En ocasiones algunas variables pueden crear ruido en los datos, pues aquellas que son redundantes o irrelevantes degradan la clasificación. La selección de variables se basa en seleccionar los atributos más revelantes para el problema, es decir, la selección de un subconjunto.
En el paquete clusterSim está la función HINoV.Mod(), permite trabajar con variables mixtas y sin clases a priori. En R, están implementados múltiples paquetes para la selección de variables, entre los cuales se encuentran: ‘FSelector’, ‘caret', ‘rpart', ‘randomForest', ‘clustvarsel’, entre otros.
Medidas de similitud
La similitud entre objetos es una medida de correspondencia, o parecido, entre objetos que van a ser agrupados. Existe un gran número de medidas de distancias y asociación que se diferencian no solo por ser de similaridad o disimilaridad, sino también por el tipo de datos, para el que han sido diseñadas, ya sean cuantitativos, cualitativos o mixtos.
El paquete stats forma parte de la librería básica de R que se instala por defecto, este contiene la función dist() que calcula diferentes medidas de distancias.
El paquete cluster contiene la función daysi() que permite seleccionar como medida de similitud la métrica de Gower para datos mixtos.
Métodos de aglomeración
Existen dos tipos básicos de agrupamientos y se distinguen por ser de naturaleza jerárquica o no jerárquica. (Johnson 2000).
Dentro de los métodos de aglomeración, el jerárquico (árboles ultramétricos) es uno de los más usado por la comunidad científica (Ferreira and Hitchcock, 2009), el cual se divide en aglomerativo y divisivo.
El paquete stats forma parte de la librería básica de R que se instala por defecto, este contiene la función hclust() que permite obtener varios análisis conglomerados jerárquicos.
Como paquete adicional para el análisis de conglomerados se encuentra el paquete cluster que amplía la gama de análisis de conglomerados, pues incluye además métodos particionales y jerárquicos divisivos.
Validación del agrupamiento
Una vez obtenido el resultado del método de aglomeración, en el análisis taxonómico es importante determinar si el conjunto de datos muestra una tendencia a formar grupos, lo cual se determina a través del Agglomerative Coefficient (AC) con la función coef.hclust() en el paquete cluster.
Por otra parte, los métodos jerárquicos imponen cierta estructura sobre los datos, y para determinar si es aceptable o si se introducen distorsiones en las relaciones originales, se usa el test de Mantel, implementado en la función mantel() del paquete vegan.
Número óptimo de conglomerados
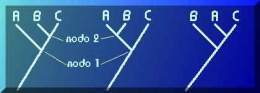
Cuando se emplean técnicas de aglomeración jerárquicas, no siempre se está interesado en la jerarquía completa, sino también en un subconjunto de particiones obtenidas a partir de ella, resultante de una determinada estructura de datos. Las particiones se obtienen cortando el dendrograma.
Las soluciones propuestas son múltiples. Por ello, surgen diversos índices de validación, algunos de los más utilizados son: Índice de Calinski-Harabasz, el índice de Davies-Bouldin, el Ancho de la Silueta (silhouette), todos implementados en el paquete clusterSim, y el índice de Dunn , implementado en el paquete clValid.
Fuentes
- Dougherty J, Kahavi R y Sahami M. Supervised and unsupervised discretization of continuous features. En Machine Learning: Proceedings of the Twelth International Conference, 1995
- Cuadras, C. M. (1981). Métodos de Análisis Multivariante. Eunibar. D.L. XII: 642
- Fawcett, T. (2003) ROC Graphs: Notes and Practical Considerations for Data Mining Researchers, Hewlett-Packard Company.
- Gower JC (1971) A general coefficient of similarity and some of its properties. Biometrics 27:857-871
- Johnson, D. E. (2000). Métodos Multivariados aplicados al análisis de datos. S.A. International Thomson Editores.
- Kohavi, R., and Provost, F. (1998), Glossary of Terms, Editorial for the Special Issue on Applications of Machine Learning and the Knowledge Discovery Process, Vol 30, No 2/3, Feb/March 1998.
- Maimon, O. and Rokach,L. (2005) Data Mining and Knowledge Discovery Handbook, Springer US, 2005.
- Podani J, Schmera D (2006) On dendrogram-based measures of functional diversity. Oikos 115:179-185
- Provost, F. and Fawcett, T. (2001). Robust classification for imprecise environments, Machine Learning 42: 203–231.
- R Core Team. 2017. R: A Language and Environment for Statistical Computing. Vienna, Austria: RFoundation for Statistical Computing. https://www.R-project.org/.
- Smith L, A Tutorial on Components Analysis, Tech. report, Universidad de Otago (Nueva Zelanda), 2002
- Sneath, P. H. A. y R. R. Sokal (1973). Numerical taxonomy. The principles and practice of numerical classification. San Francisco, California, EE.UU., W. H. Freeman and Co.
- Sokal, R. R. y P. H. A. Sneath (1963). Principies of numerical taxonomy. San Francisco y Londres, Freeman and Cía.
- Weiss, G. M., and Provost, F. (2003) Learning When Training Data are Costly: The Effect of Class Distribution on Tree Induction, JAIR 19, 315–354